近期,Chin Chem Lett在线发表了实验室王英、叶文才/华东理工大学朱彬等合作的研究论文,题目:Structure elucidation of plumerubradins A–C: correlations between 1H NMR signal patterns and structural information of [2+2]-type cyclobutane derivatives,DOI:10.1016/j.cclet.2024.110149。
[2+2]-型环丁烷天然产物(CCNPs)是一类由含双键的生源前体化合物在光或酶催化下,发生分子间或分子内的[2+2]-环加成反应而生成的次级代谢产物。因[2+2]-环加成反应具有区域选择性和立体选择性,并可经一步反应构建4个新的手性中心,故CCNPs往往具有多样的连接方式(head-to-tail或head-to-head)和复杂的立体化学结构,给该类型化合物的结构鉴定带来了挑战。长期以来,CCNPs的结构解析过程费时、费力,且容易出错。
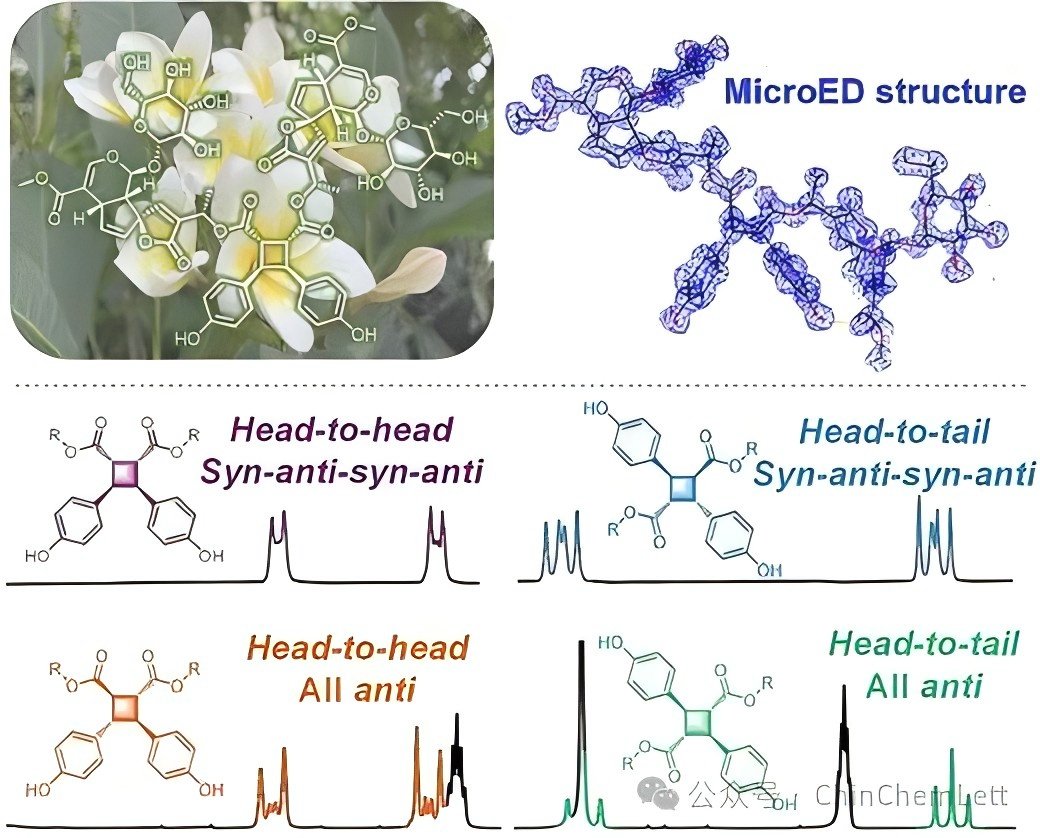
在本研究中,该团队从中药鸡蛋花(Plumeria rubra L. cv. Acutifolia)中发现了3个具有2,3-二氢吡喃[3,4-h]氧杂-螺[4,4]壬烯6/5/5三环骨架结构的鸡蛋花型环烯醚萜类化合物plumerubradins A–C。Plumerubradins A–C分别由两分子鸡蛋花型环烯醚萜苷通过[2+2]-环加成反应聚合而成,是迄今为止发现的分子量最大、结构最为复杂的环烯醚萜二聚体。由于其结构复杂,且含有多个独立的手性簇,故它们的结构解析难度极大。最终,通过综合运用波谱学、仿生半合成、化学降解、单晶X射线衍射、基于冷冻电镜的微晶电子衍射(MicroED)等方法,确定了plumerubradins A–C的结构(包括绝对构型)。其中,plumerubradin B是迄今为止利用MicroED技术解析的结构最为复杂的小分子天然产物(>1200 Da,分辨率~1 Å),说明该技术在复杂天然产物结构鉴定领域具有巨大的应用前景。进一步,该团队应用基于量子力学的1H NMR迭代全自旋分析(QM-HiFSA)方法,成功建立了环丁烷片段质子信号的峰形与其结构信息之间的关联性,并对其规律进行了总结。根据该规律,通过观察CCNPs的1H NMR图谱中环丁烷质子信号的特征峰形,即可快速确定其连接方式和立体构型。本研究为CCNPs的快速结构鉴定提供了新方法。


